查看更多

 明星产品
明星产品
 干货推荐
干货推荐
 关注我们
关注我们

扫码关注我们
了解更多信息
上期为大家介绍了自噬干货分享系列之经典自噬研究,那么本期将继续进行“自噬干货分享”,小编将带大家一起来了解下内质网自噬。

内质网是真核细胞中的一种多功能细胞器,在蛋白质折叠、修饰、脂质和激素合成、离子储存、代谢调节等多种细胞过程中发挥重要作用。当细胞受到内外因素刺激时,未折叠或错误折叠的蛋白质在内质网中大量积累,内质网的稳态就会受到破坏。此时,细胞会启动自我保护程序,即未折叠蛋白反应(unfolded protein response,UPR),增强蛋白折叠活性,降低蛋白翻译水平,加速内质网相关蛋白降解(ER-associated protein degradation,ERAD)。持续地内质网应激和未折叠蛋白反应可激活内质网自噬(ER-phagy),以维持内质网的稳态平衡。
Bernales等人在2007年首次提出了内质网自噬的概念。内质网自噬可分为(图1):大内质网自噬(是由内质网驻留受体或内质网相关受体介导,这些受体招募自噬机制,然后将内质网靶向到自噬体中,包裹内质网碎片的自噬体与溶酶体融合以降解其内容物);微内质网自噬(不需要受体或形成自噬体,内质网碎片直接被溶酶体吞噬);此外,溶酶体也可直接与内质网来源的囊泡融合并进行降解(需要内质网自噬受体介导,但不涉及自噬体的形成),这一过程也称作ERLAD(ER-to-lysosome-associated degradation)。
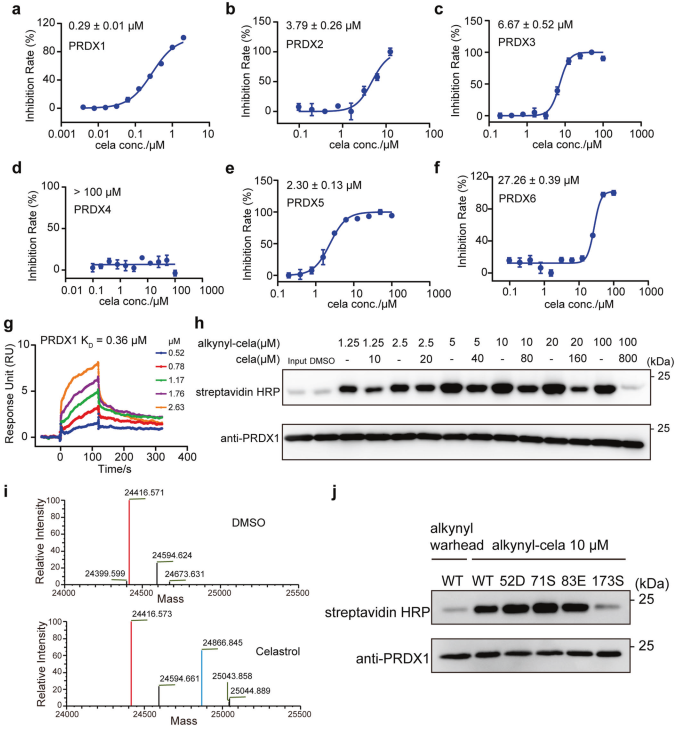
图1. 内质网自噬
本期内容主要从内质网自噬过程、内质网自噬受体、内质网自噬监测方法及应用案例来进行介绍。
1、内质网自噬过程
大内质网自噬是一个多步骤的过程:在内质网应激的诱导下(如未折叠蛋白反应、蛋白质聚集、营养缺乏和内质网结构破坏等),需要降解的内质网成分可以被特定的内质网自噬受体识别和标记。同时,细胞通过抑制mTOR或AMPK直接磷酸化Atg1/ULK1(哺乳动物中Atg1的同源物)来激活自噬体起始复合物,从而启动隔离膜的组装。类泛素蛋白Atg8/LC3/GABARAP将被招募到形成中的隔离膜上来帮助膜扩张,并识别和结合内质网自噬受体。之后,随着隔离膜逐渐扩张和闭合,与待降解的内质网子域以及内质网自噬受体形成自噬体,最终自噬体与溶酶体融合并降解底物(图2)。
图2.大内质网自噬
微内质网自噬:在酵母中,当出现内质网应激或内质网过度扩张时,一部分内质网可以形成轮状结构,然后被内陷的液泡膜吸收到液泡中进行降解。在哺乳动物细胞中,突变的I型前胶原蛋白可以被募集到 ERES(ER exit sites,内质网出口位点),COPII 外壳蛋白和自噬相关蛋白(SQSTM1、LC3 等)也可以被募集到这一位点,然后包含这些蛋白的ERES通过泛素化修饰并在微自噬等过程中被溶酶体吞没。
ERLAD:当α1-抗胰蛋白酶Z(ATZ)聚合物在内质网中积聚时,分子伴侣蛋白如CALNEXIN可以将聚集体所在的内质网亚区分离。然后,在内质网自噬受体FAM134B的协助下,在该区域形成来源于内质网的、单层膜囊泡。此外,FAM134B与LC3的结合可以促进囊泡对RAB7/LAMP1阳性的内吞溶酶体膜的对接。最终,包含聚集体的囊泡与内吞溶酶体融合而被降解。
2、内质网自噬受体
2015年Nakatogawa和Dikic团队分别在nature杂志报道了酵母和哺乳动物中第一个内质网自噬受体Atg40和FAM134B。到目前为止,哺乳动物中已经有11个内质网自噬受体被报道,FAM134B、FAM134A、FAM134C、RTN3L、CCPG1、SEC62、TEX264、ATL3、CALCOCO1、p62和CDK5RAP3(图3)。
图3.内质网自噬受体
在酵母中,内质网自噬受体通过Atg8互作基序(Atg8-interacting moti,AIM)与自噬体上的Atg8结合,促进内质网被自噬体包裹。在哺乳动物中发现了6个Atg8同源物,包括LC3 的3种亚型(LC3A, LC3B和LC3C)和γ-氨基丁酸(GABA)-A型受体相关蛋白的3种亚型(GABARAP,GABARAPL1和GABARAPL2)。与酵母中内质网自噬受体和Atg8之间的相互作用一样,哺乳动物中内质网自噬受体也通过LC3互作区(LC3-interacting
Region,LIR)与LC3和GABARAP家族蛋白结合,从而促进内质网片段被自噬体包裹。
3、内质网自噬监测方法
内质网自噬活性监测对于阐明内质网自噬的分子机制、生理和病理作用以及寻找调节内质网自噬的药物具有重要意义。
透射电子显微镜可以观察内质网自噬(图4),但无法衡量内质网自噬活性。一些内源性内质网蛋白(如RTN1、RTN3、RTN4、REEP5、CLIMP63和Trap-α)在自噬中表达水平下降,但变化通常很小。相比之下,饥饿诱导的内质网自噬受体蛋白(如CCPG1和TEX264)的降解更明显,但它们是选择性降解的,并且在内质网自噬过程中也可能被诱导转录表达,不太能真实反应内质网自噬水平。但荧光标记的报告载体是自噬研究最常用的手段,如汉恒生物自噬研究的 “王牌工具” mcherry-EGFP-LC3自噬双标病毒等,可以灵敏地衡量自噬水平。
图4. 小鼠肾组织内质网自噬透射电镜图(红色箭头表示内质网膜,黑色箭头表示自噬体膜)
基于此,汉恒生物最新推出了内质网自噬监测工具。通过将内质网定位序列与RFP-GFP荧光串联序列融合表达来构建内质网自噬报告载体ssmRFP1-EGFP-KDEL(ss:内质网信号肽序列,ER signal sequence;KDEL内质网滞留序列)(图5A)和mCherry-EGFP-RAMP4(RAMP4:即SERP1基因,Stress-associated endoplasmic reticulum protein 1,可以特异性定位到内质网膜上)(图5B)。在细胞质中,红色荧光蛋白和绿色荧光蛋白同时表达,表现为黄色;若发生内质网自噬,由于溶酶体偏酸性的环境,绿色荧光蛋白淬灭,只剩下红色荧光信号。此外,在溶酶体中,报告载体ssmRFP1-EGFP-KDEL中的mRFP1与EGFP间的linker会被剪切,因此也可通过Western Blot结果统计mRFP1/mRFP1-EGFP比值来指示内质网自噬。
图5. 汉恒内质网自噬监测工具原理图
4、应用案例
2019年东京大学研究团队为了研究TEX264是否为内质网自噬受体,利用ssmRFP1-EGFP-KDEL工具对野生型WT、FIP200-KO(自噬功能缺陷)及TEX264-KO的HeLa细胞在营养和饥饿条件下进行了内质网自噬监测,并通过在TEX264-KO的HeLa细胞中回补TEX264野生型及LIR突变型LIR4A,结合荧光成像(图6A,图6B)和Western Blot实验(图6C,图6D),证明了TEX264是一个内质网自噬受体。

图6. ssmRFP1-EGFP-KDEL工具监测HeLa细胞内质网自噬
威斯康星大学研究团队2021年的文章中为了研究Atase1或Atase2的敲除是否会促进内质网自噬,利用mCherry-EGFP-RAMP4工具在小鼠胚胎成纤维MEF细胞中监测内质网自噬。通过荧光成像(图7),统计有红色荧光斑点的细胞占比(指示自噬溶酶体),证明了Atase1敲除,而不是Atase2敲除,会促进内质网自噬。
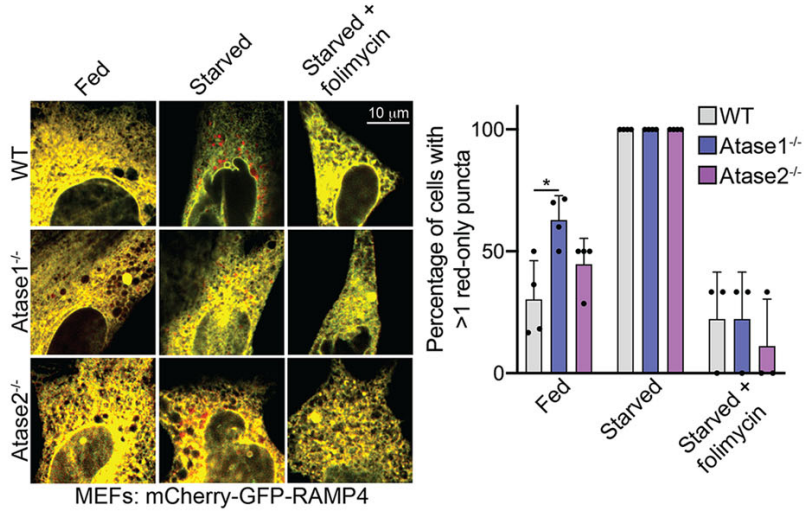
图7. mCherry-EGFP-RAMP4工具监测MEF细胞内质网自噬
本期“内质网自噬研究”的介绍就到此结束了,希望可以帮助大家了解到一定的内质网自噬基础知识,并对内质网自噬监测报告载体有一定的认识。下期我们将继续“自噬干货分享之线粒体自噬研究”,我们下期见哦。
参考文献
He L, Qian X, Cui Y. Advances in ER-Phagy and Its Diseases Relevance. Cells. 2021;10(9):2328. Published 2021 Sep 6. doi:10.3390/cells10092328
Bernales S, Schuck S, Walter P. ER-phagy: selective autophagy of the endoplasmic reticulum. Autophagy. 2007;3(3):285-287. doi:10.4161/auto.3930
Reggiori F, Molinari M. ER-phagy: mechanisms, regulation, and diseases connected to the lysosomal clearance of the endoplasmic reticulum. Physiol Rev. 2022;102(3):1393-1448. doi:10.1152/physrev.00038.2021
Chino H, Mizushima N. ER-Phagy: Quality Control and Turnover of Endoplasmic Reticulum. Trends Cell Biol. 2020;30(5):384-398. doi:10.1016/j.tcb.2020.02.001
Mochida, K., Oikawa, Y., Kimura, Y., Kirisako, H., Hirano, H., Ohsumi, Y., et al. (2015). Receptor-mediated Selective Autophagy Degrades the Endoplasmic Reticulum and the Nucleus. Nature 522, 359–362. doi:10.1038/nature14506
Khaminets, A., Heinrich, T., Mari, M., Grumati, P., Huebner, A. K., Akutsu, M., et al. (2015). Regulation of Endoplasmic Reticulum Turnover by Selective Autophagy. Nature 522, 354–358. doi:10.1038/nature14498
Mochida K, Nakatogawa H. ER-phagy: selective autophagy of the endoplasmic reticulum. EMBO Rep. 2022;23(8):e55192. doi:10.15252/embr.202255192
Li J, Gao E, Xu C, Wang H, Wei Y. ER-Phagy and Microbial Infection. Front Cell Dev Biol. 2021;9:771353. Published 2021 Nov 29. doi:10.3389/fcell.2021.771353
Chino H, Hatta T, Natsume T, Mizushima N. Intrinsically Disordered Protein TEX264 Mediates ER-phagy. Mol Cell. 2019;74(5):909-921.e6. doi:10.1016/j.molcel.2019.03.033
Liang JR, Lingeman E, Ahmed S, Corn JE. Atlastins remodel the endoplasmic reticulum for selective autophagy. J Cell Biol. 2018;217(10):3354-3367. doi:10.1083/jcb.201804185
Rigby MJ, Lawton AJ, Kaur G, et al. Endoplasmic reticulum acetyltransferases Atase1 and Atase2 differentially regulate reticulophagy, macroautophagy and cellular acetyl-CoA metabolism. Commun Biol. 2021;4(1):454. Published 2021 Apr 12. doi:10.1038/s42003-021-01992-8

查看更多

查看更多

查看更多

联系我们

返回顶部